# Instalar y cargar los paquetes necesarios
install.packages("sf")
library(sf)
# install.packages("dplyr")
library(dplyr)
# Importamos el archivo csv de GBIF con sus atributos geográficos
presencias <- read.delim("0002064-240130105604617.csv", quote = '')
# Convertimos la tabla importada a un objeto geográfico tipo sf
presencias_geo <- st_as_sf(
presencias,
coords = c("decimalLongitude", "decimalLatitude"),
crs = 4326
)
# Reproyectamos la capa creada al sistema de coordenadas EPSG:23030 (UTM)
presencias_geo_23030 <- st_transform(presencias_geo, crs = 23030)
# Creamos una malla de 250 m con la extensión y sistema de coordenadas de la capa de presencias
grid_250m <- st_make_grid(presencias_geo_23030, cellsize = c(250, 250))
# Transformamos la malla obtenida (tipo sfc) a tipo espacial sf
grid_250m_sf <- st_sf(geometry = grid_250m)
# Añadimos un campo llamado id_250 a la malla. Le incluimos valores secuenciales desde 1.
grid_250m_sf$id_250 <- seq(1, 55125, by = 1)
# Asignamos a cada punto de presencia el código del cuadrado de la malla en el que está. Unión espacial (comparar!).
presencias_x_grid <- st_join(presencias_geo_23030, left = FALSE, grid_250m_sf)
# Extraemos la tabla de atributos de la capa de puntos creada y borramos todos los campos menos los dos que nos interesan.
bio <- as.data.frame(presencias_x_grid)
bio <- bio[c('id_250', 'scientificName')]
# Calcular el número de individuos por especie y por cuadrícula (num_ind_sp_cuad): ahora en el df las cuadrículas aparecen tantas veces como especies distintas contienen y llevan asociado el número de individuos encontrados.
T_num_ind_sp_cuad <- bio %>% #create a new data table of the aggregated data
group_by(id_250, scientificName) %>% #group the calculations by id_250 & scientificName
summarize(`num_ind_sp_cuad` = length(scientificName))
# Calcular el número total de individuos por cuadrícula.
T_num_ind_cuad <- bio %>% #create a new data table of the aggregated data
group_by(id_250) %>% #group the calculations by id_250
summarize(`num_ind_cuad` = n())
# Fusionar las tablas anteriores para calcular Pi
T_num_ind_sp_cuad_mas_num_ind_cuad <- merge(T_num_ind_sp_cuad, T_num_ind_cuad)
# Calcular pi por especie y por cuadrícula.
T_num_ind_sp_cuad_mas_num_ind_cuad$pi <- T_num_ind_sp_cuad_mas_num_ind_cuad$num_ind_sp_cuad /
T_num_ind_sp_cuad_mas_num_ind_cuad$num_ind_cuad
# Calcular pi * log2(pi) por especie y por cuadrícula.
T_num_ind_sp_cuad_mas_num_ind_cuad$lnpi_pi<-log2(T_num_ind_sp_cuad_mas_num_ind_cuad$pi)*T_num_ind_sp_cuad_mas_num_ind_cuad$pi
# Calcular H por cuadrícula
T_Shannon <- T_num_ind_sp_cuad_mas_num_ind_cuad %>% #create a new data table of the aggregated data
group_by(id_250) %>% #group the calculations by id_250
summarize(`H` = sum(lnpi_pi)*(-1))
# Fusionar la tabla que tiene el índice de Shannon con la malla de cuadrículas.
grid_250m_sf <- merge(x = grid_250m_sf,
y = T_Shannon,
by.x = "id_250",
by.y = "id_250")
# Exportamos la capa de la malla obtenida a un fichero de formas para visualizarlo en QGIS.
st_write(grid_250m_sf, "Shannon_250_sierra_nevada.shp", append = FALSE)Creación de un mapa de biodiversidad de Sierra Nevada
Objetivos
Esta práctica tiene los siguientes objetivos:
- Disciplinares:
- Reconocer el concepto de biodiversidad ya estudiado en en otras ocasiones.
- Aprender un método para cuantificar la diversidad biológica: Índice de Shannon.
- Aprender a generar mapas de diversidad biológica: Mapas de distribución del índice de Shannon.
- Reconocer patrones de distribución de la diversidad en un territorio concreto.
- Identificar las causas de los patrones anteriores.
- Instrumentales:
- Afianzar nuestro conocimiento de SIG.
- Mejorar nuestro conocimiento de R.
- Aprender la forma en la que obtenemos un mapa de distribución de una variable continua a partir de datos tomados de forma discreta.
Contextualización ecológica y estructura de la sesión
Para cuantificar la diversidad biológica se pueden utilizar muchos índices. En nuestro caso usaremos el denominado índice de Shannon-Weaver, que es uno de los más robustos y comunmente utilizados. Para su cálculo se necesita la siguiente información:
- Delimitación espacial de la comunidad para la que queremos calcular el índice de diversidad.
- Listado de especies existente en esa comunidad.
- Abundancia de cada especie en dicha comunidad.

Para alcanzar los objetivos planteados, procederemos de la siguiente forma:
En primer lugar aprenderemos la mecánica del cálculo del mapa de diversidad a una escala de 250 m en el espacio protegido de Sierra Nevada (Sur de España). Es decir, asumiremos que una comunidad ecológica es una entidad de forma cuadrangular con un lado de 250 m. Esto no es así en la realidad, pero es la aproximación más fácil que tenemos a nuestro alcance.
Una vez obtenido este mapa, trataremos de estudiar los patrones de cambio espacial de dicha diversidad. También identificaremos los factores abióticos que explican dichos patrones espaciales.
Obtención de un mapa de biodiversidad: Metodología y flujo de trabajo
Como se puede observar en la presentación anterior, para calcular la diversidad de una comunidad, necesitamos dos fuentes de información:
(1) Información de distribución de especies en la zona de estudio (Sierra Nevada):
Es el primer paso fundamental porque necesitamos esta información para calcular el índice de Shannon.
Para conseguir datos de presencia de especies en Sierra Nevada usaremos una infraestructura digital denominada GBIF (Global Biodiversity Information Facility).
Se trata de un portal desde el que se tiene acceso a millones de datos de presencia de especies procedentes de colecciones biológicas (herbarios, colecciones animales, etc.) de todo el planeta.
Esta iniciativa está promovida y mantenida por multitud de países que han puesto en común toda la información de la que disponen para conocer mejor la distribución de la biodiversidad en la Tierra.
Accederemos a este portal y descargaremos toda la información de presencia de especies en Sierra Nevada. Esto nos dará una idea bastante aproximada de cómo se distribuye la diversida en esta zona. En nuestro caso, GBIF aporta una enorme cantidad de registros de presencia de especies en Sierra Nevada.
Durante la práctica visitamos el portal de GBIF y simulamos la descarga. Como este proceso puede tardar unas horas, utilizamos datos que fueron descargados anteriormente por el profesor.
Pasos para descargar manual la base de datos de especies en GBIF desde el portal web:
- Usando el navegador web entramos en gbif.org y elegimos la opción ‘Get data’ y ‘Occurrences’.
- En ‘Location’ haz clic en ‘Geometry’:
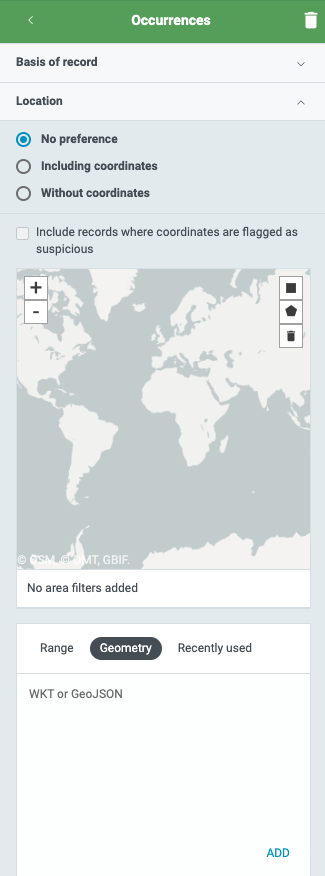
- Copia y pega el contenido del archivo con el perímetro del parque natural de Sierra Nevada. Este archivo usa el formato de coordenadas Well Known Text (WKT).
- Después haz clic en ‘ADD’. Verás la lista de ocurrencias en forma de tabla, así como en el mapa (si cambias a la pestaña ‘MAP’).
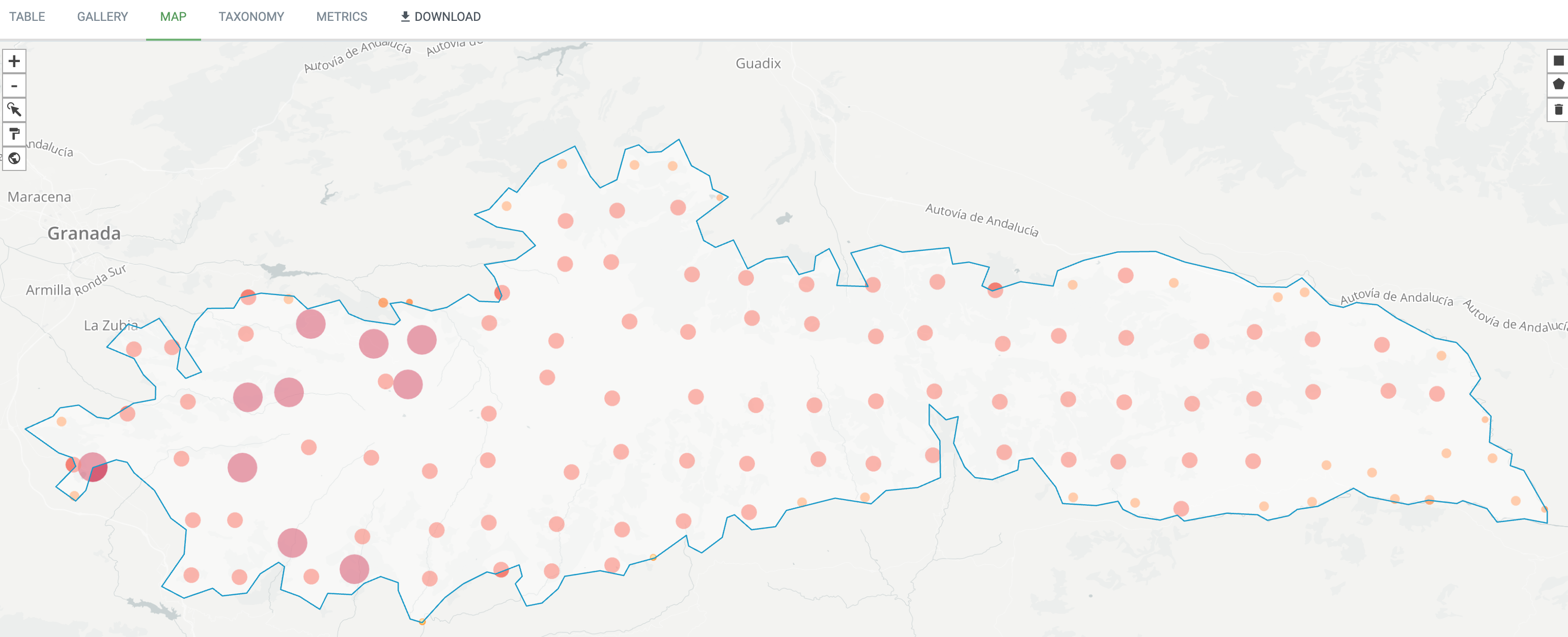
- Haz clic en la pestaña ‘DOWNLOAD’ y elige el format ‘SIMPLE’ para descargar los datos (se necesita estar registrado).
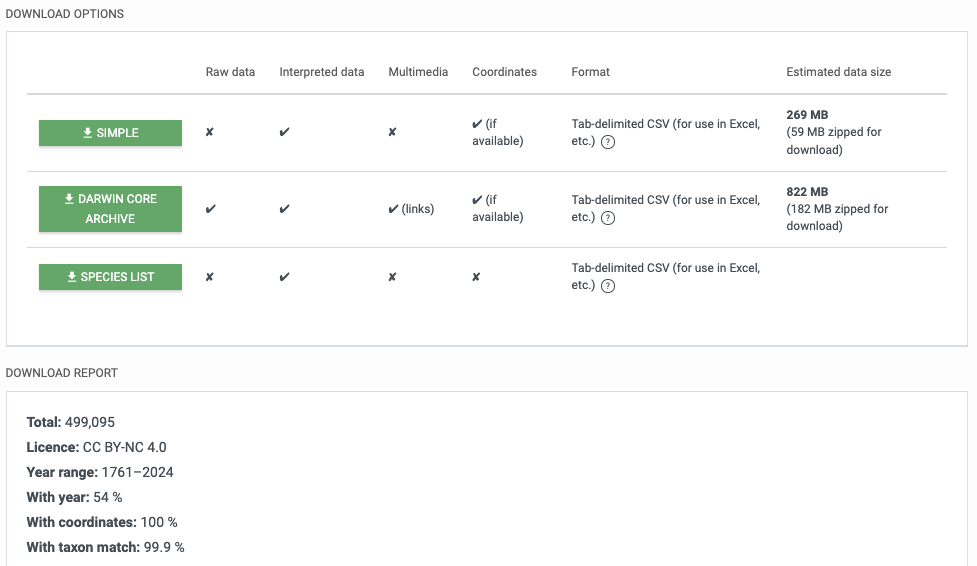
- Al descargar los datos, verás un resumen de los mismos y se generará un DOI para citarlos correctamente. Puedes descargar la cita en cualquier formato para usarla con un gestor de referencias.
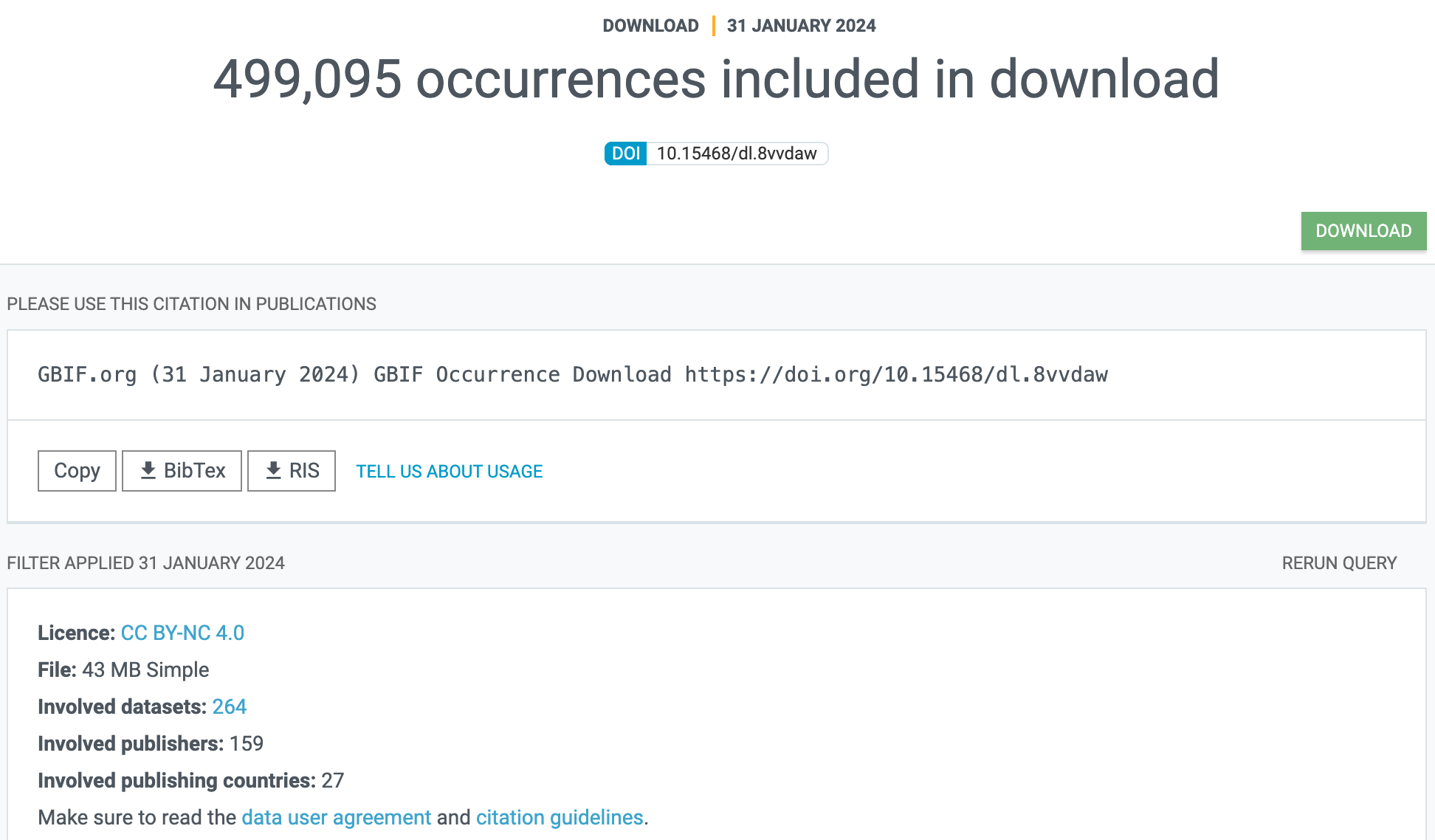
Los datos de presencia de especies tienen el siguiente “aspecto” cuando son visualizados en QGIS.
Aquí puedes descargar el archivo con los datos de presencia de especies de Sierra Nevada. Como ves más abajo, son unos cuantos miles de puntos…

(2) Distribución de las comunidades ecológicas que conforman Sierra Nevada:
Este paso es el más complejo conceptualmente, ya que las comunidades no tienen un límite espacial preciso. Es decir, están conectadas entre sí y no es fácil delimitar donde empieza una y acaba otra.
En este caso usaremos una definición artificial y arbitraria de comunidad: un cuadrado de 250 m de lado.
A partir de esta información obtendremos el índice de Shannon para cada una de las comunidades de Sierra Nevada. Es decir, usaremos los datos de presencia de cada especie que hay en cada una de las comunidades para calcular su índice de Shannon.
En la siguiente figura puedes ver la distribución de presencias de especies en unas cuantas comunidades.

Para ello seguiremos el siguiente flujo de trabajo (ya descrito a grandes rasgos en la presentación introductoria):
Importamos el archivo csv de GBIF con sus atributos geográficos.
Convertimos la tabla importada a un objeto geográfico tipo sf.
Creamos una malla de 250 m con la extensión y sistema de coordenadas de la capa de presencias.
Asignamos a cada punto de presencia el código del cuadrado de la malla en el que está. Unión espacial.
Calculamos el número de individuos por especie y por cuadrícula.
Calculamos el número total de individuos por cuadrícula.
Calculamos la proporción de individuos (pi) por especie y por cuadrícula.
Calculamos el log2 pi por especie y por cuadrícula.
Calculamos H por cuadrícula.
Exportamos la capa de la malla obtenida a un fichero de formas para visualizarlo en QGIS.
Este script de R genera un mapa del índice de Shannon en cuadrículas de 250x250 m a partir de los datos de presencias de especies existentes en GBIF.
Una vez abierto el archivo en QGIS, debemos darle una paleta de colores para visualizar los valores del índice de Shannon (columna H) correctamente:
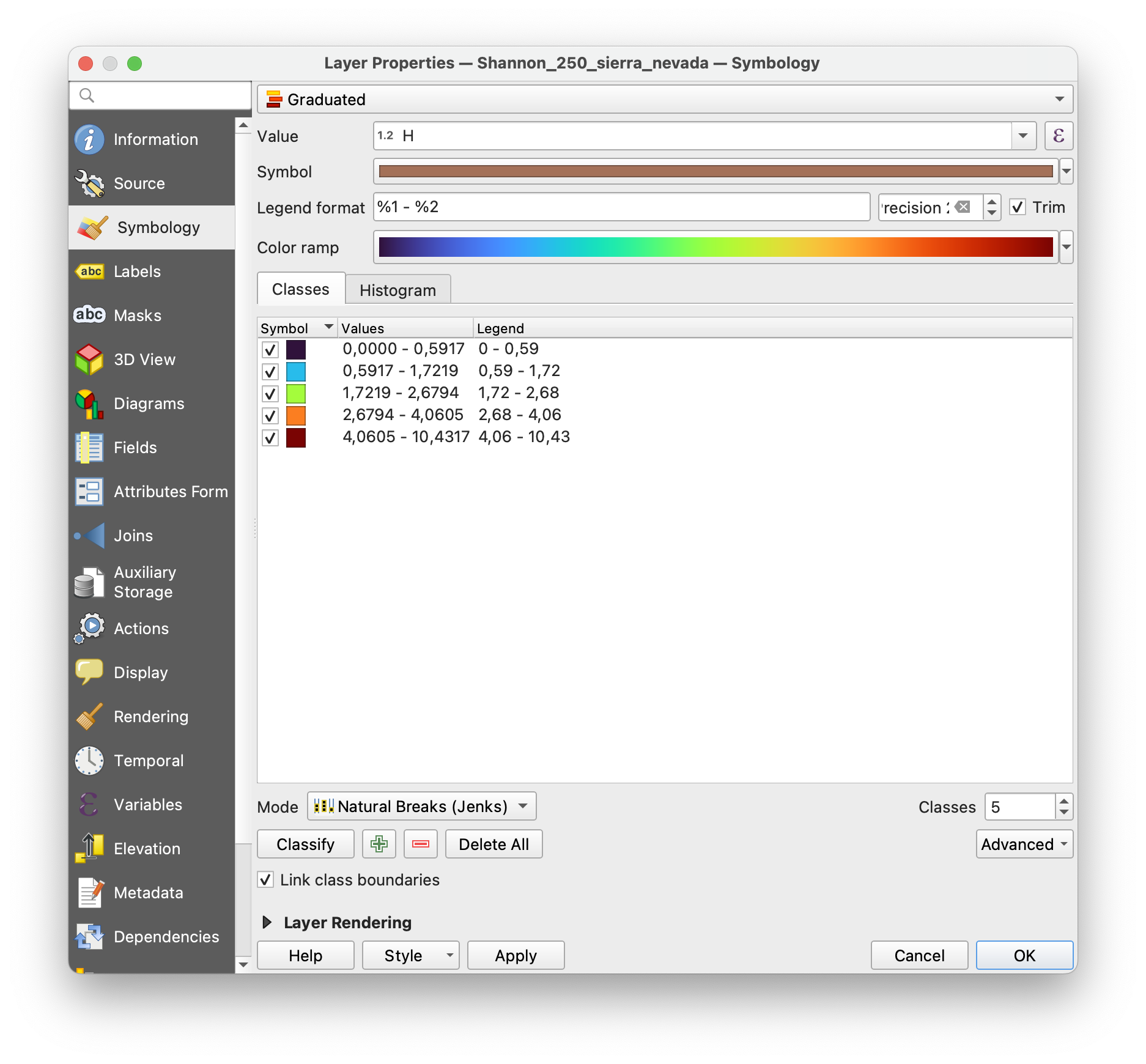
Resultados esperables
El siguiente mapa de ejemplo muestra el resultado obtenido en una práctica anterior.
Se trata de un fichero de formas vectorial en el que se ha asignado el valor del índice Shannon a cada cuadrícula de la malla de 250 m.
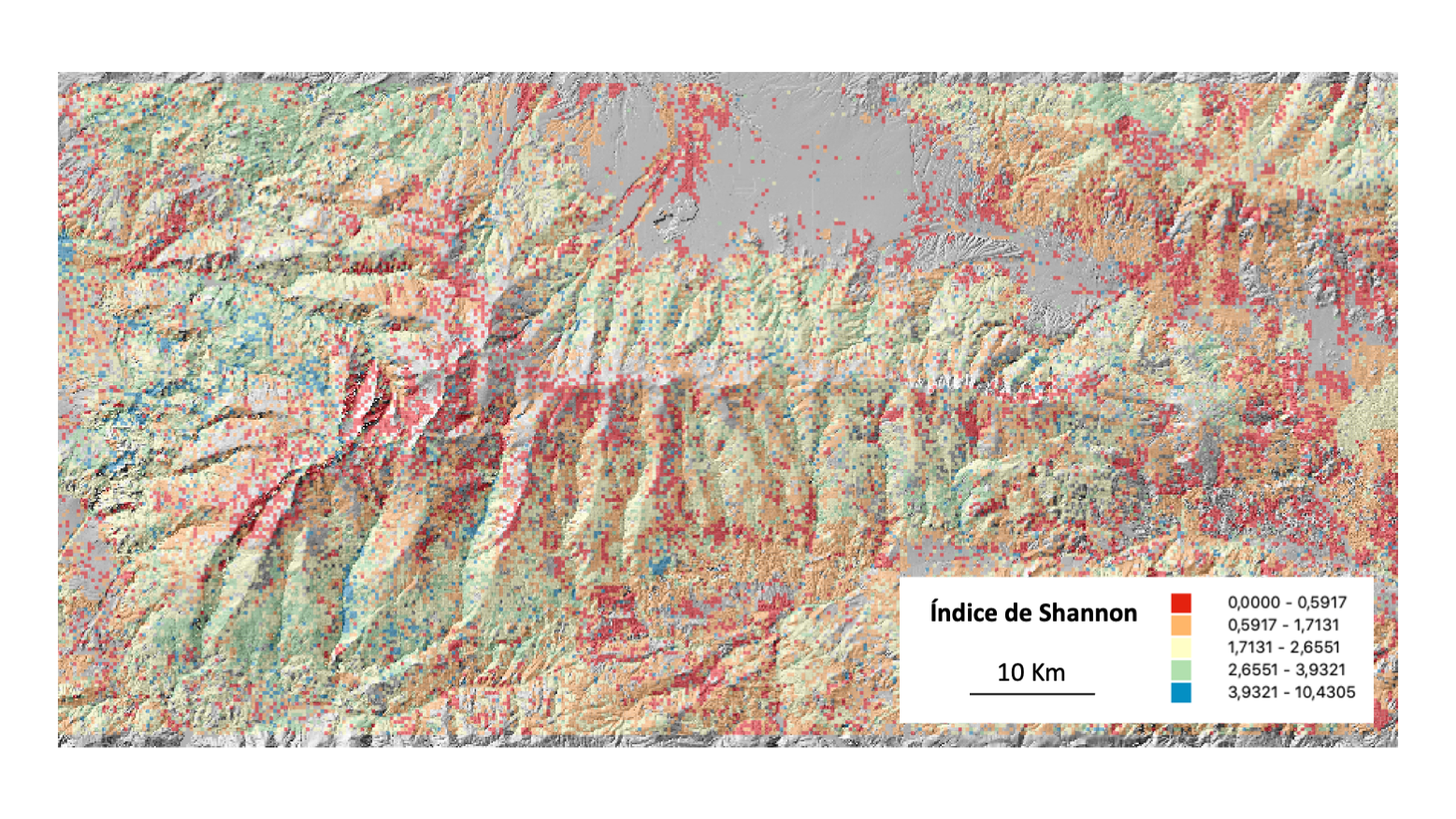
En el mapa resultante se pueden identificar varios patrones de distribución espacial de la biodiversidad en Sierra Nevada.
Durante la práctica reflexionamos sobre dichos patrones:
¿Cómo cambia la diversidad al aumentar la altitud? ¿A qué se puede deber dicho patrón?
¿Crees que se repetiría el mismo patrón en otras montañas de la Tierra?
¿Ves algún sesgo espacial en los datos de GBIF?
¿Hay algún patrón de cambio de diversidad en dirección Este-Oeste? En caso afirmativo, ¿a qué puede deberse?
Recursos adicionales
En este vídeo de Francisco Javier Bonet (UCO) podéis ver la discusión de los resultados obtenidos en una sesión anterior.
Evaluamos patrones de distribución espacial de la diversidad y tratamos de identificar las variables que explican dichos patrones.